石田恒(原子力機構)
RuvA-Holliday junction DNAの分岐点移動解析
ホリデイ構造は、DNA相同組換えの普遍的なDNA中間体である。大腸菌では、RuvA-RuvB複合体がホリデイ構造の分岐点移動を促進する。RuvAはホリデイ構造特異的DNA結合蛋白質でる。RuvBは2つの6量体がホリデイ分岐DNAと結合し、ATP加水分解エネルギーを用いてホリデイ分岐点の移動反応を触媒する。分岐点移動反応を原子レベルで理解するには、分岐点移動におけるエネルギー地形および遷移状態を解析する必要がある。そこで、ホリデイ分岐DNAに調和型ポテンシャルを加えることで分岐点移動のエネルギー地形を調べるアンブレラサンプリングシミュレーションを実行した。系は、E.Coli由来のRuvA4量体-ホリデイ分岐DNA(25塩基×4鎖)複合体の立体構造(PDBコード:1C7Y)を用いた。
結果、分岐点移動における自由エネルギー障壁は約10-15kcal/molと見積ることができた。また自由エネルギー地形とホリデイ分岐DNAの構造変化の関係を解析したところ、分岐点移動開始時には対のない13番目塩基に隣接する12-14番塩基対の水素結合が不安定化することがわかった。塩基対の水素結合が崩れた後、対を作らない塩基と隣の塩基のスタック間距離が大きくなる。遷移中間状態では、対を作らない塩基は分岐点中心にあるRuvA酸性ピンと安定化する。特に、酸性ピンはグリコシド結合角を安定な位置に維持することがわかった。以上により、RuvAがホリデイ分岐DNAの分岐点移動を促進する仕組みを構造的に理解できた。
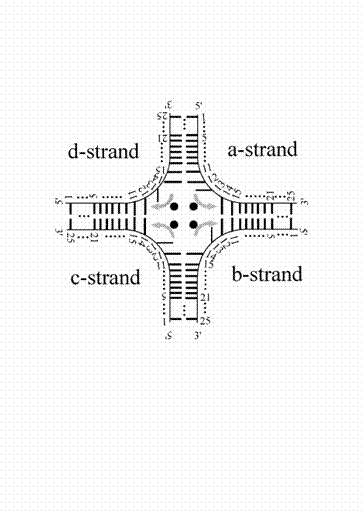
図1:ホリデイ分岐DNA(Holliday junction DNA)のモデル
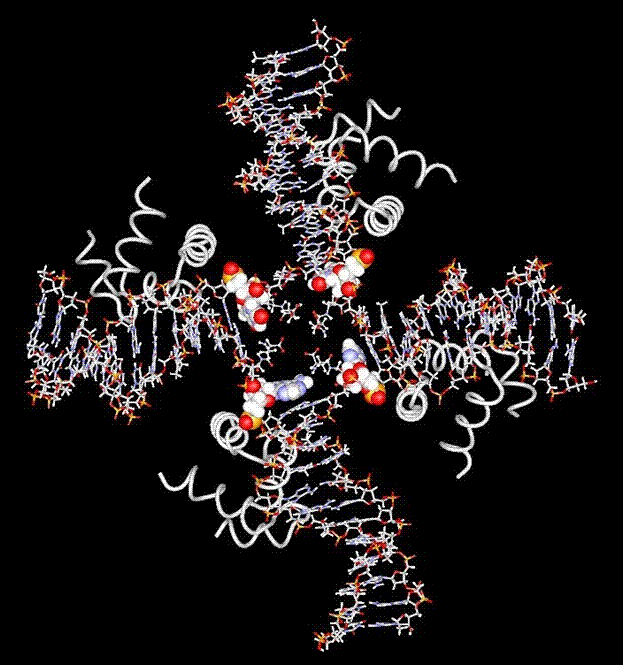
図2:ホリデイ分岐DNA(Holliday junction DNA)とRuvA(HhHモチーフのみ表示)の複合体
もとのページに戻る

